
![]() Investindo no genoma
Investindo no genoma
![]() Incentivo à pesquisa
Incentivo à pesquisa
![]() Papel da bioinformática
Papel da bioinformática
![]() Política científica
Política científica
![]() Despertando interesse
Despertando interesse
![]() A casa de Salomão - artigo
A casa de Salomão - artigo
![]() Reflexões sobre o Genoma Humano
Reflexões sobre o Genoma Humano
![]() Perfil - Andrew Simpson
Perfil - Andrew Simpson
![]() Bioética - artigo
Bioética - artigo
![]() Glossário
Glossário
| Brasil ganha capa da Nature com o seqüênciamento do genoma da bactéria Xylella. Fonte: Nature. |
|
A Xylella causa o entupimento dos vasos da laranjeira pela produção de goma xantana. Fonte Fapesp.
|
| Ramo de uma laranjeira atacada por Xylella. As manchas amareladas nas extremidades das folhas são regiões em que a bactéria se fixa e desenvolve. O ramo inteiro pode ser comprometido, junto com seus frutos. Fonte Fapesp. |
| Cana-de-açúcar, um dos principais produtos da agricultura brasileira, tem partes de seu genoma em processo de seqüenciamento. Ilustração Nelson Yoneda. |
Abrindo espaço para a genômica no Brasil
 Neste
momento em que a mídia divulga com grande destaque o primeiro rascunho
completo do genoma humano, o Brasil assiste ao evento na posição de quem
entende do assunto. Isto porque, por ação de órgãos de incentivo à ciência
e tecnologia, em especial a Fundação de Amparo à Pesquisa do Estado de
São Paulo (Fapesp), o país investiu nos últimos anos na área de genética
molecular e pôs o pé no grupo dos países que desenvolvem projetos de seqüenciamento
genético. Entrou, por sinal, com o pé direito, pois em fevereiro deste
ano tornou-se o primeiro a completar o genoma de uma bactéria que provoca
doenças em plantas (fitopatógeno), cujo nome, aliás, acabou se tornando
popular: a Xylella fastidiosa.
Neste
momento em que a mídia divulga com grande destaque o primeiro rascunho
completo do genoma humano, o Brasil assiste ao evento na posição de quem
entende do assunto. Isto porque, por ação de órgãos de incentivo à ciência
e tecnologia, em especial a Fundação de Amparo à Pesquisa do Estado de
São Paulo (Fapesp), o país investiu nos últimos anos na área de genética
molecular e pôs o pé no grupo dos países que desenvolvem projetos de seqüenciamento
genético. Entrou, por sinal, com o pé direito, pois em fevereiro deste
ano tornou-se o primeiro a completar o genoma de uma bactéria que provoca
doenças em plantas (fitopatógeno), cujo nome, aliás, acabou se tornando
popular: a Xylella fastidiosa.
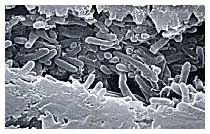 Causadora
da praga do amarelinho, a Xylella foi
escolhida pela Fapesp como organismo para seu projeto genoma piloto por
uma conjunção de fatores. Para citar os mais importantes: pelo fato de
não haver no mundo, à época, outra pesquisa de seqüenciamento de um fitopatógeno,
sua importância econômica (cerca de 30% dos laranjais paulistas são afetados
pelo amarelinho) e o tamanho de seu genoma (2,7 milhões de pares de bases,
relativamente pequeno). Prova de que a escolha foi acertada é a fala de
Peter Johnson, responsável por um dos maiores programas de financiamento
de pesquisa do departamento de Agricultura norte-americano: "Meus caros,
tenho para vocês uma boa e uma má notícia. A boa é a de que foi concluído
o seqüenciamento do primeiro fitopatógeno no mundo. A má é a de que não
fomos nós que fizemos, mas um grupo brasileiro".
Causadora
da praga do amarelinho, a Xylella foi
escolhida pela Fapesp como organismo para seu projeto genoma piloto por
uma conjunção de fatores. Para citar os mais importantes: pelo fato de
não haver no mundo, à época, outra pesquisa de seqüenciamento de um fitopatógeno,
sua importância econômica (cerca de 30% dos laranjais paulistas são afetados
pelo amarelinho) e o tamanho de seu genoma (2,7 milhões de pares de bases,
relativamente pequeno). Prova de que a escolha foi acertada é a fala de
Peter Johnson, responsável por um dos maiores programas de financiamento
de pesquisa do departamento de Agricultura norte-americano: "Meus caros,
tenho para vocês uma boa e uma má notícia. A boa é a de que foi concluído
o seqüenciamento do primeiro fitopatógeno no mundo. A má é a de que não
fomos nós que fizemos, mas um grupo brasileiro".
O reconhecimento da importância da pesquisa vem também pela publicação, este mês, do artigo científico descrevendo os resultados do projeto, na revista Nature, que aliás já havia publicado, em 1997, uma notícia dando conta do início do projeto, com destaque para o ineditismo da pesquisa com a bactéria. A publicação do artigo, como bem sabem os cientistas, é um importante fator de certificação do mérito da pesquisa e certamente fará aumentar as citações internacionais dos pesquisadores envolvidos no projeto e indiretamente da ciência brasileira. Enquanto isso, outros projetos genoma financiados pela Fapesp já estão em andamento.
 Como
conseqüência direta do Genoma
Xylella, surgiu, em 1999, o Genoma
Xylella Funcional, que busca estudar apenas os genes de caráter
patogênico da bactéria, ou seja, aqueles que mantêm relação com a praga
do amarelinho. Desta etapa participam cerca de 20 laboratórios, que no
prazo de um ano deverão apresentar seus primeiros resultados. Eles foram
selecionados entre os cerca de 100 que apresentaram propostas à Fapesp.
Como exemplo das pesquisas, há o estudo da biossíntese de goma xantana,
substância viscosa produzida pela Xylella e uma das possíveis causas
do entupimento dos vasos da laranjeira, o que causa o amarelinho.
Como
conseqüência direta do Genoma
Xylella, surgiu, em 1999, o Genoma
Xylella Funcional, que busca estudar apenas os genes de caráter
patogênico da bactéria, ou seja, aqueles que mantêm relação com a praga
do amarelinho. Desta etapa participam cerca de 20 laboratórios, que no
prazo de um ano deverão apresentar seus primeiros resultados. Eles foram
selecionados entre os cerca de 100 que apresentaram propostas à Fapesp.
Como exemplo das pesquisas, há o estudo da biossíntese de goma xantana,
substância viscosa produzida pela Xylella e uma das possíveis causas
do entupimento dos vasos da laranjeira, o que causa o amarelinho.
Outro estudo em curso, utilizando novos métodos de microscopia eletrônica, é o da adesão bacteriana no trato bucal da cigarrinha que transmite a Xylella e sua influência na propagação da doença.
 Além
dos projetos Genoma Xylella e Genoma Funcional, surgiram também
aqueles dirigidos a outros organismos. O primeiro deles foi o Genoma
Cana, iniciado em julho de 1999, com o objetivo de seqüenciar partes
escolhidas do DNA da cana-de-açúcar e identificar
cerca de 50 mil genes com características de interesse econômico, como
resistência a doenças, metabolismo da sacarose, resistência a geadas e
seca. Este projeto, que é coordenado pelo professor Paulo Arruda, da Unicamp,
conta também com um financiamento de US$ 500 mil da Copersucar
(assim como, no caso da Xylella, participou a Fundecitrus),
que disponibilizou seu Centro de Tecnologia para a realização de algumas
etapas da pesquisa. Até março deste ano foram depositados nos laboratórios
de bioinformática do projeto 68 mil partes
seqüenciadas e o objetivo é alcançar 300 mil até julho de 2003. Este prazo
será, muito provavelmente, reduzido, devido à maior velocidade de seqüenciamento
adquirida com o Genoma Xylella. Do seqüenciamento participam, além
do laboratório de bioinformática da Unicamp, coordenado pelos professores
João Meidanis e João Setúbal, também o laboratório da Universidade Federal
de Penambuco coordenado pela pesquisadora Kátia Guimarães.
Além
dos projetos Genoma Xylella e Genoma Funcional, surgiram também
aqueles dirigidos a outros organismos. O primeiro deles foi o Genoma
Cana, iniciado em julho de 1999, com o objetivo de seqüenciar partes
escolhidas do DNA da cana-de-açúcar e identificar
cerca de 50 mil genes com características de interesse econômico, como
resistência a doenças, metabolismo da sacarose, resistência a geadas e
seca. Este projeto, que é coordenado pelo professor Paulo Arruda, da Unicamp,
conta também com um financiamento de US$ 500 mil da Copersucar
(assim como, no caso da Xylella, participou a Fundecitrus),
que disponibilizou seu Centro de Tecnologia para a realização de algumas
etapas da pesquisa. Até março deste ano foram depositados nos laboratórios
de bioinformática do projeto 68 mil partes
seqüenciadas e o objetivo é alcançar 300 mil até julho de 2003. Este prazo
será, muito provavelmente, reduzido, devido à maior velocidade de seqüenciamento
adquirida com o Genoma Xylella. Do seqüenciamento participam, além
do laboratório de bioinformática da Unicamp, coordenado pelos professores
João Meidanis e João Setúbal, também o laboratório da Universidade Federal
de Penambuco coordenado pela pesquisadora Kátia Guimarães.
Quase ao mesmo tempo que o Genoma Cana surgiu o Genoma Câncer, em parceria com o Instituto Ludwig de Pesquisa do Câncer, o primeiro a entrar com um financiamento igual ao da Fapesp na pesquisa (US$ 5 milhões cada). A pesquisa, que é coordenada pelo professor Andrew Simpson, está baseada numa nova técnica de seqüenciamento, chamada ORESTES (Open Reading Frame EST), desenvolvida e patenteada pelo Ludwig. Os dados, que já resultam em 250 mil seqüências de genes, estão disponibilizados no GenBank, onde está a maior parte da informação do projeto Genoma Humano, e num site dedicado a este seqüenciamento. Deste projeto participam cinco centros de seqüenciamento, localizados na USP, Unifesp e USP/Ribeirão Preto. Espera-se chegar, até o final do ano, a 1 milhão de seqüências de genes.
Em julho deste ano, o projeto Genoma Câncer contou com um aumento de 60% no orçamento, sendo parte deste montante investido na área de bioinformática e na criação de mais dois bancos de clones. Esse projeto colocou o Brasil no segundo lugar no ranking do países que mais descreveram seqüências genéticas expressas em cânceres, ficando atrás somente do National Cancer Institute (NCI), que desenvolve pesquisa homóloga nos Estados Unidos.
Finalmente, o mais recente dos projetos genoma é o Genoma Xanthomonas, coordenado pelo professor Fernando Reinach, que procurará seqüenciar o código genético da bactéria Xanthomonas axonopodis pv citri. Ela é causadora do cancro cítrico e parente próxima da Xylella fastidiosa. Seu genoma, porém, é aproximadamente duas vezes maior que o desta última, mas deverá ser concluído em menos tempo. A comparação entre ambos gerará informações importantes sobre semelhanças e diferenças de metabolismo, ciclos de vida e interação com seu hospedeiro comum, os citros (sobretudo a laranja).
A organização do projeto inclui 12 laboratórios, sendo os dois principais da USP e da Unesp/Jaboticabal, onde serão armazenadas, processadas, comparadas e anotadas as seqüências de DNA, usando o software do projeto da Xylella. Toda a parte de bioinformática será feita pela Unicamp. Os outros laboratórios de seqüenciamento serão selecionados por um comitê da Fapesp e depois agregados à rede Onsa, formada por todos os laboratórios que participam de projetos Genoma financiados pela agência.
Desse modo, o Genoma Xylella e seus "descendentes" consolidam uma etapa importante da pesquisa científica no país e abrem campo para novos projetos...
Atualizado em 06/07/00
http://www.comciencia.br
comciencia@epub.org.br
© 2000
SBPC/Labjor
Brasil
